AlphaFold是一个AI程序,之前已经证明它可以从氨基酸序列预测蛋白质结构,现在已经与另外两个AI例程配对,即使在蛋白质结构未知的情况下,也可以提供端到端的AI药物发现过程。1这种机器学习过程的组合能够预测一种针对肝癌新靶点的新型药物样小分子,展示了人工智能如何快速准确地设计定制疗法。
“许多与疾病有关的靶点都没有已知的结构。使用结构为设计过程提供了其他方法无法比拟的优势,”公司人工智能平台负责人Petrina Kamya解释道Insilico医学该公司开发了这项技术艾伦Aspuru-Guzik加拿大多伦多大学教授,诺贝尔奖得主迈克尔·莱维特美国斯坦福大学教授。
靶蛋白的结构通常通过实验方法得到,如x射线晶体学。然而,获得这些结构可能会有困难,对于完全新颖的目标,获得x射线结构可能很耗时。
卡姆亚说:“AlphaFold是一个计算机程序,它声称已经生成了人类基因组中所有蛋白质的结构,有望解决由来已久的蛋白质折叠问题。”为了展示使用AlphaFold生成目标蛋白质结构的好处,Insilico Medicine团队将AlphaFold与他们开发的两个人工智能程序配对;熊猫组学与化学
熊猫组学可以筛选和解释大量组学数据。组学数据通常从高通量生化分析中获得,这些信息与科学文献的文本挖掘和拨款描述一起使用,以寻找可能的治疗靶点。Kamya解释说:“熊猫组学是一个目标发现引擎,它提供了对以前可能被忽视的疾病-目标关系的深入了解,有大量证据支持这种联系。”
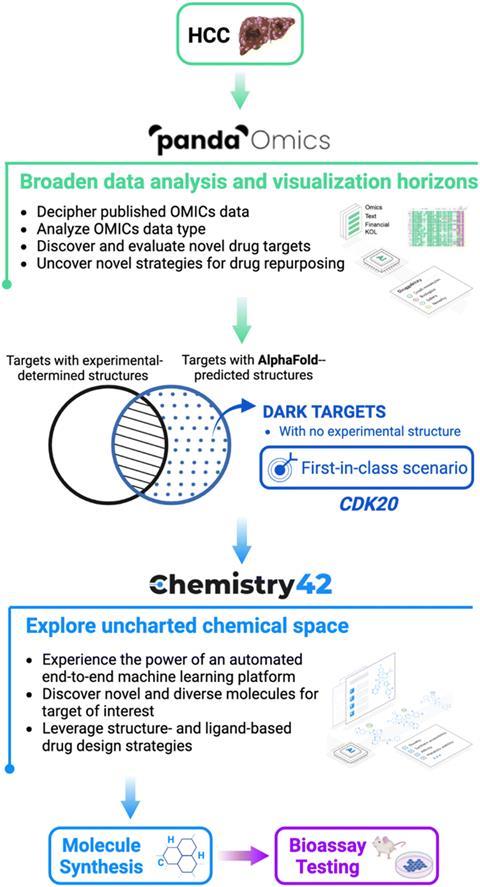
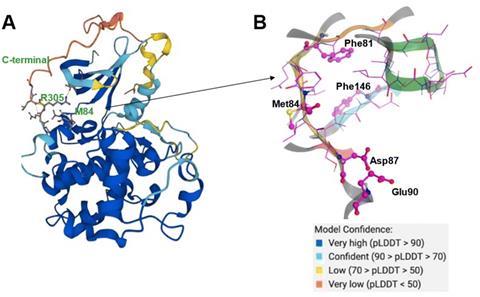
InSilico的研究团队瞄准了一种可用于治疗肝细胞癌(一种常见的肝癌)的小分子,利用熊猫组学的解释能力,发现CDK20蛋白是一种合理的治疗靶点。CDK20在肿瘤细胞系中过表达,肺癌细胞中这种蛋白质的减少与细胞增殖的减少和对放射化学治疗的敏感性增加有关。虽然从实验中无法得知蛋白质结构,但AlphaFold能够提出CDK20的可能结构。
从目标到击中的30天
接下来,该团队将AlphaFold预测的结构与生成AI Chemistry42配对,后者提出了CDK20小分子抑制剂的结合位点。通过分析alphafold预测的蛋白质结构,Chemistry42提出了150Å的浅结合袋3..通过人工智能的生成程序对这个口袋进行进一步研究,发现它将合理的化学结构、部分和官能团编织在一起,从而提出一系列配体。这些配体是围绕CDK20内结合袋的3D形状、可用的体积以及关键原子和蛋白质残基的空间排列而设计的。人工智能提出了54种潜在的抑制剂,该团队合成了其中7种。这个过程从目标选择到第一次命中仅用了30天,随后chemisty42的改进发现了更有效的命中分子,在实验测试中表现出抗癌活性。
“与药物研发活动所需的时间和投资相比,这种完全由人工智能驱动的方法已经显示出巨大的优势,并显示了完全由人工智能预测和从头生成化学结构驱动的药物发现的范式转变,”评论道巴勃罗Carbonell)他是西班牙瓦伦西亚大学的计算生物学家。
对于Insilico Medicine的团队来说,下一个目标之一是将端到端人工智能药物发现整合到机器人实验室中,例如他们在中国苏州的新设施。Aspuru-Guzik解释说,自主运行的实验室可以将人工智能主导的药物发现与自动化反应和药物配方结合起来2由机器学习提出的加速过程。
Aspuru-Guzik补充道:“我认为这一趋势的下一个自然演变是公开药物研发。”“应该有联邦政府资助的非营利性机构来研究被忽视的疾病。随着这些工具变得越来越便宜,越来越民主,为什么不为此建立一个联邦机构呢?”
参考文献
1楼任等,化学。科学。, 2023, doi:10.1039 / d2sc05709c
2 P班尼根等,Commun性质。, 2023, doi:10.1038 / s41467 - 022 - 35343 - w









1读者的评论